|
2021年6月9日,团队在《Nature Communication》在线发表题为“Whole-genome microsynteny-based phylogeny of angiosperms”的论文

系统发育重建对研究生物各类群间亲缘关系与进化演变至关重要,也是探索重要性状的起源与演化、提出相应科学猜想的框架基石。虽然当前大量的全基因组数据、转录组数据极大地丰富了可供分析的信息位点,依然有一些分支关系饱受争议。当前系统发育重建主要基于多序列比对的方法,然而受进化历程中古多倍化事件、不完全谱系分拣、杂交渐渗等因素的影响,标记基因选取和系统发育关系的真实重建面临诸多挑战。共线性(synteny),即基因沿染色体的排列次序(gene order) 反映的是基因组的结构信息,是独立于基因序列信息之外的一层信息,也可以用来进行系统发育重建,而且相较于序列变异,得出的结果可能更为可靠。
该研究基于前期开发的共线性网络分析方法(Zhao et al, Plant Cell, 2017; Zhao & Schranz, PNAS, 2019),开发了Syn-MRL的方法进行系统发育重建,将共线性网络数据库中全部基因的共线性基因簇信息(synteny cluster)转化为二元矩阵表示(binary matrix representation),最后用最大似然法(maximum likelihood)构建物种树(图1)。
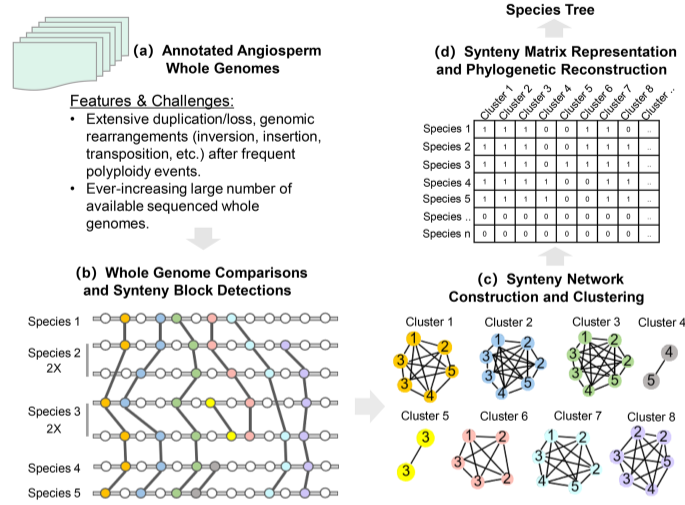
图1 基于全基因共线性的系统发育重建
该研究首先用模拟数据(simulation)和真实数据集校验(benchmarking datasets)对方法进行了评估,结果表明Syn-MRL具有很高的准确度和可靠性(图2)。之后,该研究使用123个高质量被子植物基因组,用Syn-MRL和基于序列比对的方法(两套标记基因*三种方法)分别构建物种树,并将结果与APG(Angiosperm Phylogeny Group)IV、1KP(One Thousand Plant Transcriptome Initiative)进行了综合比较。
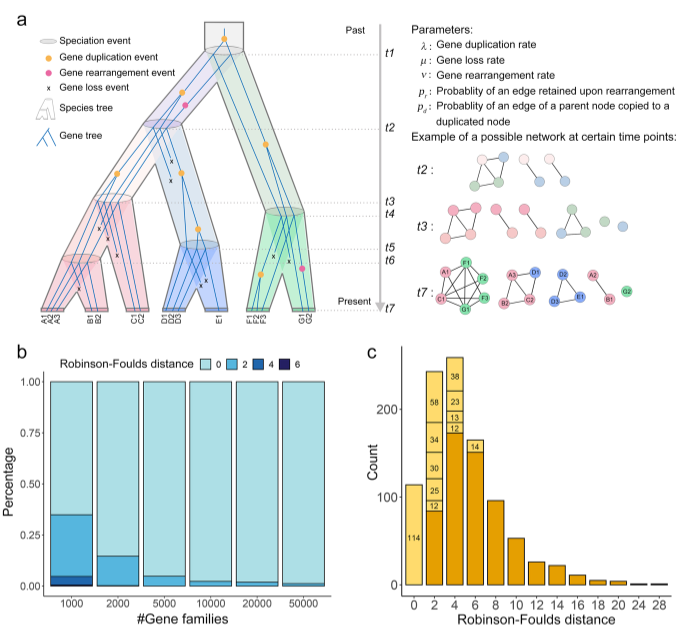
图2 Syn-MRL 方法数据模拟的参数和结果
结果表明,基于共线性的进化树与基于序列比对和权威参考的进化树高度一致,符合当前人们对主要类群间亲缘关系的共识。同时,对于一些长久以来有争议的类群,从基因组结构变异(共线性特征)的角度提出了具有较高支持率的分支结构,例如木兰类植物和单子叶植物形成姊妹群,葡萄目在真双子叶植物基部、发生位置在山龙眼目之后等等(图3)。
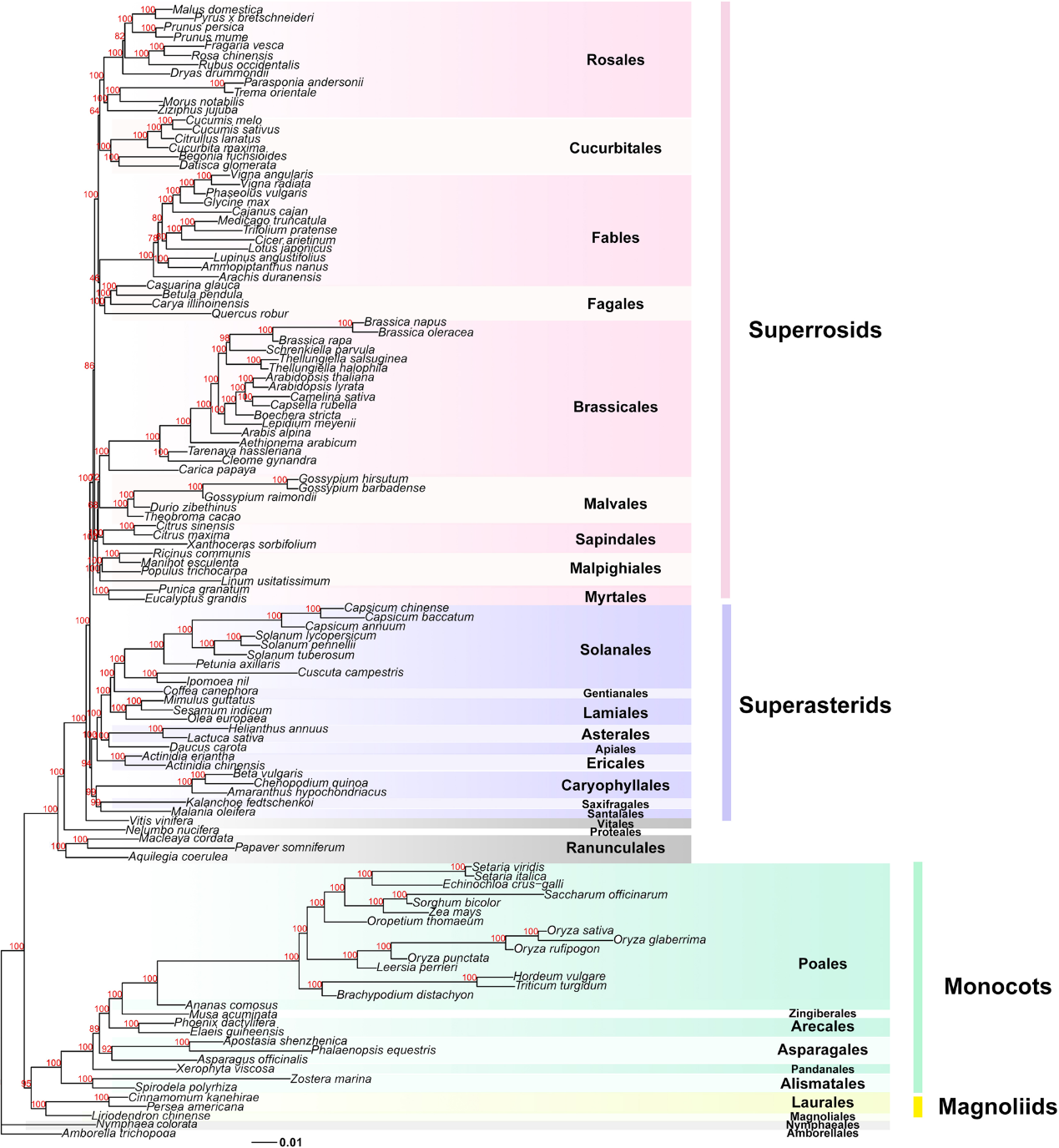
图3 基于Syn-MRL的系统发育重建
该研究也对比分析了在杂交渐渗背景下传统方法存在的潜在问题,这些结果对一些长期有争议的系统发生关系提供了新的数据层面上的证据,也为系统发育的研究提供了新的方法和思考。
南京农业大学园艺学院薛佳宇副教授、根特大学Arthur Zwaenepoel、李臻博士、高树民博士和荷兰瓦赫宁根大学Eric Schranz教授也为该研究做出重要贡献。
论文链接:https://www.nature.com/articles/s41467-021-23665-0
| 
